Bijna iedereen heeft wel eens een antibioticumkuur gehad om een infectie te bestrijden. Antibiotica worden voornamelijk door schimmels gemaakt en zijn het verdedigingsmechanisme van deze schimmels tegen bacteriën. De hedendaagse antibiotica zijn meestal varianten van natuurlijke isolaten, ook wel semi-synthetische antibiotica genoemd. Er zijn twee redenen om natuurlijke antibiotica te veranderen. Ten eerste is door veelvuldig en verkeerd gebruik van antibiotica resistentie opgetreden tegen de natuurlijke isolaten. Zo is de MRSA bacterie (Methicilline resistente Staphylococcus aureus), die regelmatig in ziekenhuizen de kop opsteekt, alleen nog maar gevoelig voor één bekend antibioticum. Daarnaast zijn de natuurlijke antibiotica niet zo stabiel en is het spectrum van bacteriën die ze kunnen doden beperkt.
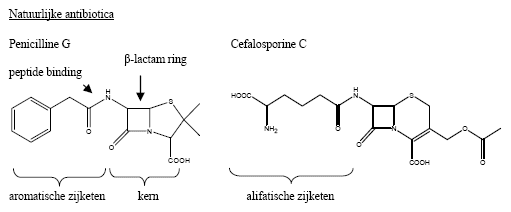
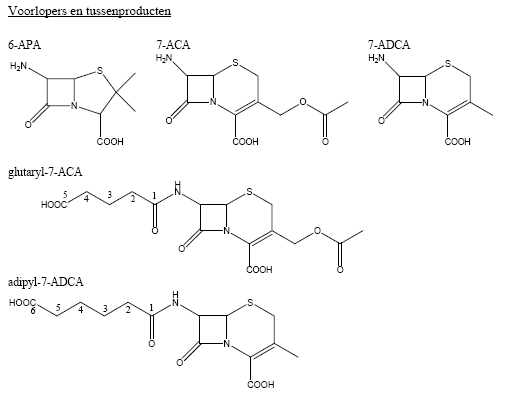
Op basis van de chemische structuur zijn verschillende soorten antibiotica te onderscheiden en de antibiotica die op penicilline G lijken, worden de β-lactam antibiotica genoemd (Figuur 1). Penicilline G bestaat uit een kern van een 5-ring plus een 4-ring, een β-lactam groep, met daaraan gekoppeld een zijketen. Een ander, in Nederland minder bekend, β-lactam antibioticum is cefalosporine (Figuur 1). Deze verbinding heeft een 6-ring in plaats van een 5-ring in de kern, waardoor het antibioticum in staat is andere bacteriën te doden dan penicillines. De natuurlijke zijketen van het antibioticum cefalosporine C is niet een aromatische ring, zoals bij penicilline G, maar een lange alifatische keten van zes C-atomen met een carboxyl groep en een amino-groep ook wel aminoadipyl genaamd. Veranderingen in de zijketens veranderen de stabiliteit en werking van β-lactam antibiotica. Zo heeft de zijketen van ampicilline in vergelijking tot penicilline G slechts één extra hydroxyl groep, maar dit antibioticum kan daardoor wel oraal geven worden, terwijl penicilline G direct door het maagzuur wordt afgebroken. Het vervangen van de zijketen van β-lactam antibiotica wordt al jaren industrieel toegepast door eerst de bestaande zijketen bij de peptide binding af te knippen (deacyleren) en er een nieuwe zijketen op dezelfde plek aan te zetten (acyleren). Dit proces wordt tegenwoordig door enzymen gedaan, omdat enzymen heel specifiek bepaalde chemische reacties kunnen katalyseren. Het enzym, dat gebruikt wordt bij het maken van penicilline-achtige antibiotica, wordt penicilline acylase genoemd. Dit enzym heeft een voorkeur voor aromatische zijketens en kan daarom niet de alifatische zijketen van cefalosporines afknippen. Voor deze antibiotica zijn cefalosporine acylases nodig. Helaas zijn er in de natuur nog steeds geen cefalosporine acylases gevonden die de zijketen van cefalosporine C accepteren. Daarom wordt de zijketen van cefalosporine C in twee stappen afgeknipt. In de eerste stap wordt de zijketen één C atoom en één aminogroep korter gemaakt en in de tweede stap wordt de resterende (glutaryl)zijketen door het enzym glutaryl acylase afgesplitst. Dit enzym kan een alifatische zijketen van vijf C-atomen heel goed afsplitsen, zoals de zijketen van glutaryl-7-ACA (Figuur 1) en ook een zijketen met zes C-atomen, zoals adipyl-7-ADCA, wordt langzaam door dit enzym afgesplitst. De aminoadipyl-zijketen van cefalosporine C wordt echter niet door dit enzym herkend en dus ook bijna niet afgeknipt (Figuur 1). Enigszins verwarrend is de naamgeving. Hoewel het enzym nauwelijks activiteit op cefalosporine C vertoont, wordt dit enzym toch tot de familie der cefalosporine acylases gerekend.
Semi-synthetische cefalosporines kunnen ook uit penicillines worden gemaakt door de 5-ring te vergroten naar een 6-ring en daarna de zijketen eraf te knippen met penicilline acylase. Deze methode is een chemisch ingewikkeld proces dat veel energie kost en waarbij erg veel (chemisch) afval ontstaat. Aangezien dit erg slecht voor het milieu is en afvalverwerking steeds duurder wordt, is men al jaren op zoek naar nieuwe manieren om semi-synthetische cefalosporines te maken. Een stap in de goede richting was de ontdekking van een enzym dat de 5-ring van penicilline om kan zetten in een 6-ring. Helaas bleek dit enzym niet te werken als de penicilline zijketen aan de ring zat. Wel bleek het enzym te werken als je de schimmel in plaats van de aromatische zijketen een alifatische zijketen met zes C-atomen gaf. Dit heeft geleid tot een genetische gemodificeerde schimmel, die adipyl-7-ADCA in plaats van penicilline G produceert. De zijketen van deze stof kan door een cefalosporine acylase langzaam worden afgeknipt. Door de reactie omstandigheden te veranderen kan de reactie versneld worden, waardoor het proces economisch haalbaar wordt. De snelheid is echter nog steeds niet te vergelijken met het penicilline acylase en daarom werd het onderzoek opgestart, dat in dit proefschrift is beschreven. Het doel was om een bestaand cefalosporine acylase zodanig te veranderen dat het de zijketen van adipyl-7-ADCA en misschien zelfs die van cefalosporine C even efficiënt kan afknippen als het originele enzym de zijketen van glutaryl-7-ACA kan hydrolyseren. Voor dit onderzoek werd het glutaryl acylase van de bacterie Pseudomonas SY-77 gekozen om mee te beginnen. Het gen, dat voor dit enzym codeert, kan in de veelgebruikte gastheer Escherichia coli genetisch worden veranderd, waarna de mutante enzymen door deze bacterie worden geproduceerd.
Er zijn verschillende manieren om enzymen te veranderen. Wij hebben ervoor gekozen om de tactiek van Moeder Natuur af te kijken. Tijdens de evolutie zijn steeds nieuwe enzymen en enzymen met nieuwe activiteiten ontstaan, doordat er continu kleine mutaties en recombinaties optraden. Veel mutaties kunnen een enzym zodanig veranderen dat het een andere activiteit krijgt. Recombinatie is het opnieuw rangschikken van DNA en treedt vooral op bij de productie van ei- en zaadcellen, waarbij de chromosomen van vader en moeder uit elkaar getrokken worden. Soms zijn deze chromosomen zo verstrengeld dat breuken optreden en genen van vader en moeder worden uitgewisseld. Daarnaast zorgen veranderende omstandigheden in de natuur ervoor dat er steeds nieuwe enzymen nodig zijn, waardoor je een survival-of-the-fittest krijgt.
In het lab kan de evolutie versneld nagebootst worden door genen van enzymen te mutageniseren en te recombineren en daarna de gewenste activiteit van één van de mutanten te selecteren. Omdat de onderzoeker het proces heel gericht een bepaalde kant op stuurt, wordt deze methode wel gerichte evolutie genoemd (in het engels directed evolution). In dit proces zijn twee dingen cruciaal, de manier waarop de mutanten gemaakt worden en de methode waarmee het beste enzym uit de groep geselecteerd wordt. Een verzameling van zoveel mogelijk verschillende mutanten wordt een mutantenbank genoemd. Om de perfecte mutantenbank te maken, moet je met genoeg diversiteit beginnen. Deze diversiteit kun je bereiken door het DNA te mutageniseren of door combinaties van natuurlijke varianten te gebruiken. Er zijn vele wegen die naar Rome leiden, en zo zijn er ook veel verschillende technieken ontwikkeld om goede mutantenbanken te maken. Iedere techniek heeft zijn eigen voor- en nadelen en het hangt van meerdere factoren af welke methode het meest geschikt is voor het doel dat je met een bepaald onderzoek wilt bereiken.
Het tweede belangrijke deel van het gerichte evolutieproces is het vinden van de beste mutant. Dit kan op twee manieren, door middel van selectie of screening. In een selectiemethode wordt ervoor gezorgd dat de gewenste enzymactiviteit de overleving van de bacterie bevordert. Dit lijkt dus het meest op de selectie zoals die in de natuur plaatsvindt. Het voordeel van deze methode is dat alleen de beste mutanten overleven, terwijl bacteriën die enzymen produceren die helemaal niet meer actief zijn, dood gaan. Helaas kan niet voor iedere enzymactiviteit een selectiemethode ontwikkeld worden. Dan is een screeningsmethode het alternatief. In een screening worden alle mutanten individueel getest op de gewenste activiteit. Het nadeel is dus dat je ook alle slechtere en inactieve mutanten moet testen Door het inzetten van robots kunnen steeds meer screeningsactiviteiten snel worden uitgevoerd. Dit wordt High Throughput Screening genoemd.
Het bovenstaande is een samenvatting van hoofdstuk 1, waarin een algemene inleiding wordt gegeven over het enzym en de technieken, die tot nu toe zijn ontwikkeld om gerichte evolutie uit te voeren. Bij de start van dit onderzoek waren al verschillende gerichte evolutie experimenten beschreven en uitgevoerd op modelsystemen met makkelijk meetbare enzymactiviteiten. Dit is noodzakelijk om een nieuwe techniek te ontwikkelen, maar levert vaak geen praktisch bruikbaar enzym op. Daarom is besloten om de waarde van deze technieken te testen met een industrieel relevant enzym, dat meteen toegepast kan worden in de productie van semi-synthetische antibiotica. Dit betekende echter wel dat er een goede selectie methode opgezet moest worden en een manier om de activiteit van de nieuwe enzymen te meten.



Figuur 1. Structuren van veelvoorkomende antibiotica en hun voorlopers.
In hoofdstuk 2 wordt een mutagenese methode ontwikkeld om mutanten van het SY-77 glutaryl acylase te maken. Het acylase bestaat uit twee delen (subunits), een kleine α- en een grotere β-subunit. De α-subunit was al eerder gemutageniseerd, daarom hebben we ons op de β-subunit gericht. Uit praktische overwegingen hebben we dit deel van het gen in vijf overlappende delen opgesplitst en elk apart gemutageniseerd. Hiervoor is de random mutagenese strategie error-prone PCR (polymerase kettingreactie) gebruikt. Met deze methode kopieer je DNA in een reageerbuis met behulp van een DNA polymerase. Door tijdens de reactie een aantal omstandigheden niet optimaal te houden, gaat het polymerase fouten maken. Wij hebben de reactie zodanig verstoord dat er gemiddeld 1-2 fouten per deel werden ingebouwd. Hierdoor ontstaat een mutantenbank met allemaal varianten van het enzym. De varianten in de vijf mutantenbanken zijn vervolgens geselecteerd op hun activiteit om een adipyl-zijketen af te splitsen. Hiertoe zijn de enzymen geproduceerd in E. coli DH10B, een bacterie die zelf geen leucine kan maken. Leucine is een aminozuur dat in bijna alle eiwitten voorkomt en is dus noodzakelijk voor de groei van de bacterie. Door nu de bacteriën te laten groeien op een medium met leucine gekoppeld aan de adipyl-zijketen, zullen alleen bacteriën die deze zijketen kunnen afsplitsen, en daarmee leucine vrijmaken, kunnen groeien. We hebben de snelst groeiende kolonies opgepikt en verder geanalyseerd. Daarbij is gekeken of de mutante enzymen in staat waren om het gewenste substraat adipyl-7-ADCA en het oorspronkelijke substraat glutaryl-7-ACA te hydrolyseren. De ratio van deze twee activiteiten werd gebruikt als maat voor de verandering van de enzymactiviteit. De DNA-sequentie, en daarmee de aminozuurvolgorde van de mutanten, werd bepaald en het bleek dat de meest voorkomende mutanten een ander aminozuur hadden op positie 266 of 375. Vijf mutanten hadden een verbeterde adipyl-7-ADCA/glutaryl-7-ACA hydrolyse ratio en deze enzymen werden opgezuiverd uit de bacterie om de enzymatische parameters preciezer te bepalen. Het bleek dat het enzym SY-77N266H, waarin het aminozuur asparagine (N) op positie 266 vervangen is door een histidine (H), het substraat adipyl-7-ADCA bijna 10 keer beter kan hydrolyseren dan het originele SY-77 acylase. De mutant SY-77F375L (fenylalanine op positie 375 naar een leucine), die de beste adipyl/glutaryl ratio had, bleek niet te zijn verbeterd in het afsplitsen van de zijketen van adipyl-7-ADCA, maar verslechterd in de hydrolyse van glutaryl-7-ACA. Het is dus erg belangrijk om de precieze parameters van het gezuiverde enzym te bepalen en niet alleen naar de ratio van activiteit op het ‘oude’ en ‘nieuwe’ substraat te kijken.
Door de aard van de error-prone PCR techniek kan er slechts een gering aantal DNA mutaties en daarmee ook slechts een deel van alle mogelijke aminozuren ingebouwd worden. Omdat uit de resultaten leek dat posities 266 en 375 belangrijk zijn voor de substraatspecificiteit van het enzym, is ervoor gekozen om deze posities volledig te randomiseren met behulp van de zogenaamde site-saturation mutagenesetechniek. Dit wordt in hoofdstuk 3 en 4 beschreven. Alle mogelijke varianten zijn opgegroeid om het enzym te zuiveren. Het bleek dat slechts vier mutanten, die een aminozuurverandering in positie 266 hadden, niet gemaakt konden worden. Van de overige mutanten werd de activiteit voor de hydrolyse van glutaryl-7-ACA, adipyl-7-ADCA en cefalosporine C bepaald. Elke mutant bleek zijn eigen profiel te hebben ten opzichte van deze drie substraten. Dit bevestigt dat de aminozuren op deze twee posities erg belangrijk zijn voor de substraatspecificiteit van het enzym. Bijna alle mutanten zijn verslechterd in de hydrolyse van glutaryl-7-ACA. Dit is logisch, omdat het originele enzym dit substraat heel goed afbreekt. De mutanten die adipyl-7-ADCA het beste kunnen hydrolyseren zijn SY-77N266M (asparagine naar methionine) en SY-77F375C (fenylalanine naar cysteine). De mutant SY-77N266Q (asparagine naar glutamine) bleek cefalosporine C drie keer beter om te zetten dan het originele enzym. Dit is een veelbelovend resultaat, ook al is dit altijd nog een factor 100 lager dan de hydrolyse-activiteit ten opzichte van glutaryl-7-ACA. Al deze aminozuurveranderingen kunnen alleen maar ontstaan door twee of zelfs drie DNA-mutaties en hebben dus een zeer kleine kans aanwezig te zijn in een error-prone PCR mutantenbank. Hieruit blijkt dus dat de gevolgde strategie een goede manier is om een gericht evolutie experiment te doen: eerst zoek je de posities in een enzym die belangrijk zijn voor substraatspecificiteit en daarna verander je de aminozuren op die posities in alle 19 andere aminozuren om te bepalen welk aminozuur op die plek tot de hoogste gewenste activiteit leidt.
Ondanks het feit dat er al behoorlijke verbeteringen van de adipyl-7-ADCA hydrolyseactiviteit was gevonden, is deze activiteit nog steeds niet zo goed als de activiteit van het originele enzym ten opzichte van glutaryl-7-ACA. Het leek ons daarom interessant om te proberen deze activiteit nog verder te verbeteren door meervoudige mutanten te maken. In hoofdstuk 5 worden dan ook zowel rationele als random mutagenese strategieën beschreven om de beste meervoudige mutant te vinden. Eerst werd de beste mutatie in de β-subunit, histidine in plaats van tyrosine op positie 178 (Y178H), gecombineerd met een aantal van de beste mutaties in de β-subunit. Hieruit bleek dat de combinatie van mutatie Y178H met F375L (fenylalanine naar leucine) een betere hydrolyse-activiteit ten opzichte van de enkele mutanten gaf, terwijl de andere combinaties niet tot een verbetering voerden. Hieruit blijkt dat effecten van mutaties niet altijd additioneel zijn, oftewel één plus één is niet altijd twee.
Omdat combinaties van mutaties niet altijd een voorspelbaar resultaat opleveren, werden de belangrijke posities op een willekeurige manier gecombineerd. Alle mogelijke 20 aminozuren op positie 266 (uit hoofdstuk 3) werden gecombineerd met alle mogelijke aminozuren op positie 375 (uit hoofdstuk 4). Daarnaast werd deze mutantenbank ook nog gecombineerd met mutatie Y178H in de α-subunit. Omdat deze banken uit 20 x 20 = 400 verschillende combinaties bestaan, hebben we de beste mutanten eruit gepikt door de bacteriën te selecteren met de selectiemethode beschreven in hoofdstuk 2. Er werden twee soorten platen gebruikt, één met adipyl-leucine en één met aminoadipyl-leucine. Helaas groeiden er bijna geen kolonies op de platen met aminoadipyl-leucine en leidde deze selectie niet tot nieuwe mutanten. Van de kolonies die van de adipyl-leucine platen waren opgepikt bleken er maar vier verschillende mutanten te zijn die een verbeterde ratio van adipyl-7-ADCA/glutaryl-7-ACA hydrolyse te hebben. Deze mutanten werden gezuiverd en de enzymatische parameters werden bepaald. De mutant met de hoogste adipyl-7-ADCA hydrolyse activiteit, SY-77Y178H+N266M, bleek een combinatie te zijn van de beste enkelvoudige mutanten uit de α- en β-subunit en had dus ook rationeel gemaakt kunnen worden. Cefalosporine C bleek beter omgezet te worden door de enkele mutant SY-77Y178H dan door elk van de vier geselecteerde mutanten. Dit is niet geheel onverwacht, omdat deze mutanten op adipyl-leucine waren geselecteerd en niet op aminoadipyl-leucine.
Als laatste poging om een verbeterd acylase te maken, werden de vijf belangrijke aminozuren voor substraatspecificiteit totaal gerandomiseerd en gecombineerd. Hiertoe werd het stukje gen waarin de aminozuren Tyr178, Tyr231, Arg255 en Asn266 (tyrosine, arginine en asparagine) aanwezig zijn gemaakt met behulp van synthetische stukjes DNA, oligonucleotiden genoemd. De basen die voor deze aminozuren coderen werden gerandomiseerd, zodat codons voor elk aminozuur gevormd kunnen worden. Alle benodigde oligonucleotiden werden aan elkaar geplakt en het zo ontstane genfragment werd vermenigvuldigd met behulp van PCR. Alhoewel dit in theorie heel makkelijk lijkt, was er wel wat optimalisatie nodig om een mutantenbank van 3*106 mutanten te krijgen, die alle mogelijke combinaties bevat. Het synthetische stuk DNA werd teruggeplaatst in een bank van het SY-77 gen waarin op positie 375 alle 20 aminozuren aanwezig waren (uit hoofdstuk 4). Helaas was deze laatste stap niet erg succesvol, waardoor slechts 0.13% van de totaal mogelijke grootte voor de mutantenbank werd verkregen. Hopend op wat geluk (andere onderzoekers hebben al eens verbeterde mutanten gevonden met een bank die slechts 0.01% van de totaal mogelijke populatie omvatte), werd de bank toch op selectieve platen met aminoadipylleucine geselecteerd. Helaas leverde dit geen verbeterde mutanten op. Het experiment, en vooral de laatste stap, zal dan ook geoptimaliseerd moeten worden, maar dit kon niet meer in dit proefschrift worden beschreven.